
全球首个! 襄禾资本投资企业「华深智药」完成单条序列蛋白质结构预测最后一块拼图 | 襄禾Family
近日,襄禾资本投资企业「华深智药」正式对外宣布,在AI和生命科学结合领域实现一个重大的突破,在蛋白质结构预测方面开发出全新技术OmegaFold。OmegaFold,这是全球范围内首个解决了已有计算机预测三维结构的模式,仅从单条蛋白序列就能预测出蛋白质的3D结构的算法,这在计算生物史上是一个程碑式的进展。
作为深耕于AI+新药开发领域的重要参与者,华深智药是由清华大学智能产业研究院(AIR)孵化,致力于使用AI重构药物开发流程来提高新药研发速度和效率的企业。襄禾资本于2021年领投华深智药天使轮融资,并进一步加码A轮融资。此后华深智药发展迅速,凭借行业领先的核心技术和独有的商业模式,先后获得高瓴创投、五源资本、高榕资本等多个知名机构投资的追捧。
据了解,几十年来,人们一直在尝试着在计算机内模拟和预测蛋白质三维结构折叠,相应的算法也层出不穷。在OmegaFold之前,由谷歌旗下DeepMind发布的最佳的方法AlphaFold2和RoseTTAFold是从一个蛋白质的进化历史中提取氨基酸的共进化信息,并根据这种共进化信息预测蛋白质的三维结构。也就是说,如果要预测人体中的某个蛋白质的三维结构,目前的算法需要提前在数据库中搜索与该蛋白质在进化当中拥有共同祖先(同源)的其他物种中的类似的蛋白质的序列。但是,很多蛋白质缺乏这种同源信息,例如,近年来出现了大量人工设计的蛋白质药物和工业合成用酶,都是在生物进化历史中从未出现过的。因此,AlphaFold2和RoseTTAFold在这一大类蛋白质上也是束手无策。在6月19日,清华大学智能产业研究院(AIR)高级访问教授、华深智药创始人彭健博士在社交平台上分享了Helixon团队的重大突破性科学进展。“蛋白质在体内进行折叠并不需要同源序列的存在,也不需要知道任何进化信息,我们的团队也一直坚信蛋白质的三维结构可以仅仅从他的序列信息当中预测,本着科学研究的第一性原理,我们团队终于开发出了仅从单条蛋白序列就能预测出3D结构的算法OmegaFold。”

OmegaFold的出现让人类从此不依赖一个蛋白质必须在进化当中留有印记,就可以获知他的三维结构信息并进一步推断他的生物功能。这将大大拓宽了人类设计蛋白质的搜索空间和效率。在最近的全球持续蛋白质结构预测竞赛CAMEO和全球蛋白质结构预测竞赛CASP13/14的数据集上,OmegaFold从单条序列预测的结构分值远远超过了AlphaFold2以及RoseTTAFold,并且整体达到了或超越了AlphaFold2和RoseTTAFold的MSA版本的预测精度。
除了在CASP和CAMEO这些传统数据集上,Helxion团队还将视角聚焦到了AlphaFold2和RoseTTAFold的软肋:两类众所周知的缺乏蛋白质同源进化信息的蛋白质,一类是抗体蛋白质,另一类是所谓的孤儿蛋白质。其中抗体是人体免疫系统在抗原刺激下应激产生的,原理上就不会留有进化信息,一直是传统蛋白质三维结构预测软件的盲区。OmegaFold软件在这两类蛋白质上,尤其是抗体的关键功能区的结构预测上,取得了突破性的进展。这将给整个大分子制药领域带来前所未有的变革和机会。

和传统的方法比较,OmegaFold利用了更少的信息,但是在多项指标上却可以赶超AlphaFold2和RoseTTAFold,这主要归功于OmegaFold在概念上的突破以及蛋白质序列大模型以及几何深度学习模型的创新应用。Helixon团队发现,传统的端到端的三维结构预测方法,在很多情况下,无法为每个原子都生成“合法”的坐标。在欧几里得空间中,预测出来的原子之间的距离连最起码的三角不等式都无法满足。Helixon团队采用了一种基于几何信息的深度学习模型,该模型一方面根据序列信息预测原子坐标,另一方面还学习预测原子之间在三维空间中的距离,同时,通过不断迭代的方法使得这些预测出来的距离满足基本的几何定律。这种简单的对于直觉的刻画,也直接替代了AlphaFold2和RoseTTAFold所需要的同源进化信息。
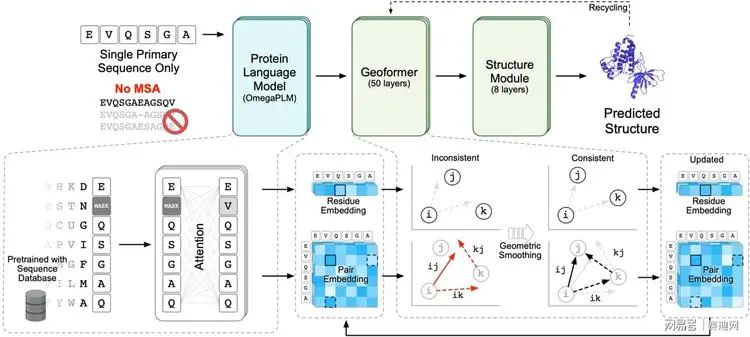
Helixon团队还同时发现,仅仅通过氨基酸序列来预测三维结构,不仅减少了同源序列所带来的噪音,还能够提高训练和预测的计算速度,使得蛋白质结构可以在数秒内被预测完毕。

OmegaFold的出现补全了蛋白质三维结构预测的最后一块拼图,它有望和AlphaFold2一起携手彻底解决这一困扰人类长达半个世纪的难题。彭健博士相信这仅仅是一个开始。在他看来,除了已有的结构预测,未来蛋白间相互作用、以及蛋白质设计手段都将迅速被一批新的方法所颠覆。





